Sciences & Société
Soutenance de thèse : Margot SARACCO
Adaptation membranaire chez les Archées hyperthermophiles: Rôle des têtes polaires dans la stabilité des membranes
Doctorante : Margot SARACCO
Laboratoire INSA : MAP - MICROBIOLOGIE, ADAPTATION ET PATHOGENIE
École doctorale : n°341 E2M2 - Evolution Ecosystèmes Microbiologie Modélisation
Parmi les trois domaines du vivant, les Archées se distinguent par la structure singulière de leurs lipides membranaires, constitués de deux chaînes polyisoprénoïdes liées au glycérol par des liaisons éther, contrairement aux chaînes d'acides gras estérifiées des Bactéries et des Eucaryotes. Cette composition unique confère à leurs membranes une stabilité exceptionnelle, leur permettant de survivre dans des environnements extrêmes. Malgré leur rôle central, la diversité et les voies de biosynthèse des lipides polaires archéens demeurent encore largement méconnues. Dans ce contexte, cette thèse explore l'organisation membranaire de Pyrococcus furiosus, une Archée hyperthermophile. Historiquement, sa membrane était décrite comme composée majoritairement de phosphatidylinositol-diéther (PI-DGD). Cependant, grâce à une approche combinant génétique et analyses lipidomiques, il a été démontré que le PI-DGD n'est en réalité qu'un constituant mineur de la membrane de P. furiosus, co-éluant avec deux autres lipides: le phosphatidylglucose (PGlc-DGD) et le phosphatidylmannose. Ces résultats remettent en question les annotations historiques des lipides et soulignent la nécessité de valider expérimentalement ces identifications. Ils révèlent également des erreurs d'annotation de gènes supposés impliqués dans la biosynthèse de PI-DGD. Grâce à cette approche génétique et notamment via la construction de souches mutantes de P. furiosus, une inhibition partielle de la biosynthèse du lipide majoritaire PGlc-DGD a été obtenue. Cela a permis d'identifier cinq tétraéthers portant des têtes polaires héxoses et/ou N-acétylées. Une nouvelle structure lipidique, hexaéther, a également été identifiée, combinant des chaines diéthers et tétraéthers. Le répertoire lipidique de P. furiosus comprend désormais 7 diéthers et 11 tétraéthers. En tenant compte des degrés d'insaturation, du nombre de cycles et de la diversité des têtes polaires, plus de 300 structures lipidiques sont potentiellement synthétisées, décrivant une complexité lipidique proche des Eucaryotes. Afin d'évaluer la pertinence biologique de cette diversité lipidique, des expériences in vivo et in vitro ont été réalisées. Face à une baisse de température ou à une augmentation de la salinité, P. furiosus remodèle les chaînes isoprénoïdes et les têtes polaires de ses lipides membranaires. Des ajustements du nombre de cycles dans les tétraéthers et du taux d'insaturation des diéthers ont été observés. Fait notable, la proportion de diéthers reste constante à 10 %, indépendamment des conditions, un comportement original parmi les Thermococcales. Pour en explorer la fonction, des membranes ont été reconstruites avec des proportions variables de diéthers et tétraéthers. L'analyse par diffraction de neutrons a révélé que les membranes mixtes présentent une stabilité supérieure à celles constituées d'un seul type de lipide, illustrant la complémentarité fonctionnelle entre les chaînes flexibles des diéthers et les structures rigides des tétraéthers. Au-delà de la structure des chaînes isoprénoïdes, les têtes polaires jouent également un rôle crucial dans le comportement membranaire. L'acétylation des hexoses favorise la stabilité membranaire, tandis que des modifications de la composition en hexose modulent les propriétés membranaires, probablement via des variations de leur capacité à former des liaisons hydrogène. Ces observations suggèrent que la nature des têtes polaires est un facteur clé, mais encore sous-estimé, de l'adaptation membranaire chez les Archées. En conclusion, cette thèse met en lumière le rôle central de la diversité lipidique dans l'adaptation et la stabilité des membranes archéennes. Elle souligne l'importance des têtes polaires dans l'organisation et la résilience membranaire, et fournit un cadre solide pour de futures recherches biochimiques, biophysiques et biotechnologiques, notamment dans le développement de membranes bio-inspirées pour la biologie de synthèse.
Informations complémentaires
-
Amphithéâtre Émilie du Châtelet, Bibliothèque Marie Curie, INSA Lyon, 31 avenue Jean Capelle, 69621 Villeurbanne
Derniers évènements
Tous les évènements
Recherche
Des bactéries pour recycler les batteries usagées ?
Pour réussir sa transition vers une mobilité décarbonée, la France dépend encore de l’importation de matériaux critiques, comme le lithium et le cobalt. Face à cette dépendance qui sous-tend de nombreux enjeux socio-économiques et écologiques, la recherche d’alternatives pour sécuriser ces ressources se fait urgente.
Parmi l’une des solutions viables : le recyclage des objets riches en métaux grâce à un procédé de biolixiviation. À partir de bactéries, cette technique consiste à faire « digérer » des gisements secondaires, des déchets riches en métaux, par des organismes vivants, pour séparer les composés. Si la création d’une filière dédiée pourrait mettre plusieurs décennies à voir le jour, des initiatives sont en cours de développement dans les laboratoires. C’est le cas au laboratoire DEEP1 de l’INSA Lyon, où Bruno Sialve, enseignant-chercheur et Gabrielle Deslandes, doctorante, explorent la voie biologique pour récolter les métaux des batteries en fin de vie de véhicules électriques.
De la nécessité d’une solution durable pour le recyclage des batteries
L’électrification est l’un des leviers qui participent à la transition d’une mobilité décarbonée. Seulement, pour faire rouler des véhicules à faible émission, il faut produire des batteries. Et pour produire des batteries, il faut des éléments particuliers tels que le lithium, le cobalt ou le nickel. Derrière ces éléments se cachent des impacts environnementaux et des enjeux sociaux très lourds : pollution des sols et des eaux, violations des droits de l'homme dans certains pays contrôlant la majeure partie de ces matières et risques géopolitiques majeurs. Dans ce contexte, le recyclage de ces métaux devient donc une priorité pour limiter cette dépendance, et réduire les coûts écologiques de leur extraction. « Par ailleurs, une directive européenne va inciter d’une part à récupérer le lithium dans les batteries usagées (50 % d’ici à la fin de 2027 et 80 % d’ici à la fin de 2031) et d'autre part à inciter les fabricants à utiliser au moins 6% de lithium recyclé dans les nouvelles batteries. Dès 2031, ce règlement s’appliquera aussi à d’autres métaux stratégiques, avec des taux requis de 16 % pour le cobalt, 85 % pour le plomb et 6 % pour le nickel 2, une raison de plus pour accélérer notre capacité à recycler ces objets », met en contexte Bruno Sialve.
Zepf V, Reller A, Rennie C, Ashfield M & Simmons J, BP (2014): Materials critical to the energy industry.
Différents processus de recyclage déjà à l’épreuve
Plusieurs méthodes de recyclage sont déjà connues et éprouvées. D’abord, la pyrométallurgie qui consiste à fondre les résidus de batteries pour séparer les métaux par fusion. Bien que cette technique soit couramment utilisée, elle est extrêmement énergivore et peu précise, ce qui peut entraîner la perte de certains métaux rares et une grande consommation de ressources. L’hydrométallurgie propose une alternative chimique pour séparer les métaux. Moins gourmande en énergie que sa voisine, elle nécessite tout de même l'usage de produits chimiques potentiellement polluants, impliquant des risques environnementaux élevés et un traitement rigoureux des déchets générés. « Et puis, il existe l’approche biologique, qui emprunte ses grands principes à l’hydrométallurgie : pour produire les acides et reproduire les réactions d’acidolyse, ce sont des populations de bactéries qui remplacent les usines de production de produits chimiques », explique le chercheur du laboratoire DEEP.
Mettre les bactéries au boulot
La biolixiviation, c’est le terme désigné pour ce processus de recyclage par voie biologique, n’est pas nouvelle. À grande échelle, l’activité a vu le jour dans les années 1990, sur le continent africain : solution économique, elle était plus adaptée aux minerais locaux alors difficiles à traiter par des méthodes de lixiviation classiques. « Pour comprendre le mécanisme de la biolixiviation, il faut se représenter des métaux piégés dans une matrice. L’objectif est de les en libérer, en générant un lixiviat enrichi en métaux solubilisés. Pour le produire, on sélectionne des populations microbiennes, selon leurs propriétés connues. Ces organismes vont créer des conditions physico-chimiques précises, pour transformer la matière », indique le chercheur.

Essai de biolixiviation en cours à partir de « black mass », un déchet de batterie (crédits : Bruno Sialve)
Mais pour créer les conditions adéquates à ces écosystèmes complexes, il est nécessaire de les sélectionner et de les adapter. « Il s’agit de « forcer » le microbiote à exprimer des fonctions biologiques données, comme par exemple, oxyder le fer ou produire de l’acide sulfurique. Ensuite, on les met en contact avec nos déchets de batteries, pour observer l’évolution et l’adaptation de la population : est-elle encore vivante ? A-t-elle travaillé efficacement en solubilisant les métaux, etc. ? Et puis, on adapte en fonction des résultats. »
Une fois le jus de métaux généré, plusieurs techniques permettent d’extraire les précieux éléments, pour être à leur tour, réutilisés. « Nos travaux de recherche s’attachent à répondre au premier enjeu du recyclage de ces batteries, c’est-à-dire, séparer les métaux. La biolixiviation est une technique éprouvée par l’industrie minière qui, une fois maîtrisée sur certains types de gisement comme les déchets de batteries de voitures électriques, pourra potentiellement être appliquée à grande échelle, car la filière n’existe pas encore. On pourrait également imaginer l’appliquer à d’autres gisements de déchets riches en métaux ou encore liés à la transition énergétique, comme les éléments de stockage ou les aimants des éoliennes. »
Dans un contexte où la France cherche à renforcer la diversification de ses sources d’approvisionnement en éléments stratégiques, ce processus pourrait ouvrir une voie prometteuse, appliquée à l’échelle industrielle. Au carrefour de l’écologie microbienne et de la biotechnologie, le recyclage par la voie biologique s’intègrerait dans une économie circulaire, limitant la multiplication d’extractions de métaux d’intérêt, et les problématiques environnementales associées.
[1] Déchets Eaux Environnement Pollutions
[2] Source : Règles de durabilité pour les batteries et les déchets de batteries

Sciences & Société
[Escape Game] Enquête microbiologie : sauverez-vous votre burger - frites ? - FDS2024
Vous aurez une heure pour résoudre des énigmes qui vous guideront à travers le monde de la microbiologie pour identifier et lutter contre le coupable !
Une animation proposée dans le cadre de la fête de la Science 2024.
Intervenant : Unité Microbiologie Adaptation Pathogénie UMR5240
Un champ de patates est ravagé par une infection microbienne : à vous d'identifier le coupable et le remède pour que les frites ne disparaissent pas !
L'image représente une bactérie déguisée en chef cuisinier qui tient dans les mains des frites Un temps d'échange sur les métiers de la recherche sera proposé à la suite de l'escape game.
Informations complémentaires
- scd.animation@insa-lyon.fr
- https://bibliotheque.insa-lyon.fr/cms/articleview/id/7061
-
Bibliothèque Marie Curie
Derniers évènements
Tous les évènements
Sciences & Société
Soutenance de thèse : Typhaine BRUAL
Unraveling virulence regulation in pectinolytic bacteria: Insights from ArcZ and RsmC. How do these molecular maestros shape virulence in the Dickeya genus?
Doctorante : Typhaine BRUAL
Laboratoire INSA : MAP
Ecole doctorale : ED341 : Évolution, Écosystèmes, Microbiologie, Modélisation
Pectinolytic bacteria of the genus Dickeya thrive in diverse ecological niches including water, soil and plants, adapting to complex and ever-changing environments shaped by various biotic and abiotic interactions.
In this thesis, we investigated the mechanisms that regulate the virulence of the genus Dickeya, in particular D. dadantii and D. solani.
Our main findings concern post-transcriptional regulation exerted by the sRNA ArcZ and post-translational regulation modulated by the protein RsmC.
ArcZ is a key player in D. solani adaptation, regulating motility according to environmental conditions and enhancing virulence during plant infection. In addition, ArcZ plays a critical role in acid resistance.
RsmC, in turn, is involved in the regulation of motility and has a complex role in virulence. Our results suggest novel interactions for RsmC and open perspectives for studying other undocumented functions.
In a word, our study reveals how regulators such as ArcZ and RsmC orchestrate bacterial responses to a dynamic environment. These findings highlight the adaptability of Dickeya species and underscore the importance of ecological context in the study of bacterial behavior.
Informations complémentaires
-
Amphithéâtre Caullery, Bâtiment Darwin C, INSA-Lyon (Villeurbanne)
Derniers évènements
Tous les évènements
Sciences & Société
Soutenance de thèse : Maïwenn PINEAU
Régulation globale de la transcription bactérienne par le surenroulement de l’ADN
Doctorante : Maïwenn PINEAU
Laboratoire INSA : MAP
Ecole doctorale : ED 341 : E2M2 – Evolution, Ecosystèmes, Microbiologie, Modélisation
Chez les bactéries, le chromosome est situé dans le cytoplasme, sous une forme très compacte. Cette compaction résulte notamment du surenroulement de l'ADN (SC), c'est à dire de la déformation torsionnelle de la double-hélice de l’ADN. Selon les conditions environnementales rencontrées par les bactéries, le niveau de SC peut varier. Ce niveau est principalement contrôlé par la gyrase, qui augmente le niveau de SC, et la topoisomérase I, qui relâche l'ADN. La régulation du niveau de SC est très importante car le SC est un régulateur de l'expression des gènes. L'objectif de ma thèse est de caractériser la régulation globale de la transcription bactérienne par le SC. Nous avons obtenu le premier transcriptome d'une bactérie à Gram négatif à une inhibition de sa topoisomérase I avec un antibiotique. Nous avons mis une nouvelle fois en évidence une régulation globale et complexe de la transcription par le SC et nous avons découvert que la réponse des gènes à une variation de SC dépend du niveau d'expression et du contexte génomique des gènes, de la direction de la variation de SC et du contexte physiologique de la bactérie. J’ai ensuite développé un package Python facilitant les analyses statistiques reproductibles de données expérimentales (ChIP-Seq, RNA-Seq…) et d'annotations dans le cadre de l'étude de l'expression d'un génome bactérien selon ses propriétés spatiales. Avec ce package j’ai pu exploiter des données publiées sur la fixation de la topoisomérase I et de la gyrase le long du chromosome. En analysant ces données qui donnent un aperçu du niveau de SC local, j'ai pu, d’une part, étudier l'effet de la transcription sur le niveau de SC à une échelle de 10 à 20 kb autour des unités de transcription et, d’autre part, observer une organisation méconnue du chromosome en domaines de 100 kb environ.
Informations complémentaires
-
Salle de conférence de la bibliothèque universitaire des sciences, Université Lyon 1 (Villeurbanne)
Derniers évènements
Tous les évènements
Sciences & Société
Soutenance de l'Habilitation à Diriger des Recherches en sciences : Xavier MARSAULT
Architecture générative inspirée
Chercheur : Xavier MARSAULT
Laboratoire INSA : MAP (UMR 3495)
Composition du jury :
Rapporteurs :
- Marjorie MUSY
- Stéphane MERILLOU
- Vincent COURBOULAY
Jury :
|
Civilité |
Nom et Prénom |
Grade/Qualité |
Etablissement |
|---|---|---|---|
|
Mme |
MUSY Marjorie |
DR / rapporteure |
CEREMA |
|
Mr |
MERILLOU Stéphane |
PR / rapporteur |
Université Limoges |
|
Mr |
COURBOULAY Vincent |
MCF / rapporteur |
Université La Rochelle |
|
Mme |
SERVIERES Myriam |
PR / examinatrice |
CENTRALE Nantes |
|
Mr |
SHOENAUER Marc |
DR / examinateur |
INRIA |
|
Mr |
PETIT Jean-Marc |
PR / examinateur |
INSA de Lyon |
|
Mr |
GALIN Eric |
PR / examinateur |
Université Lyon I |
|
Mr |
DE LUCA Livio |
DR / examinateur |
CNRS |
|
Mr |
GESQUIERE Gilles |
PR / invité |
Université Lyon I |
|
Mr |
TERRACOL Pascal |
PR / invité |
ENSA Paris Val de Seine |
Informations complémentaires
-
ENSAL (Vaulx en Velin)
Derniers évènements
Tous les évènements
Sciences & Société
Soutenance de l'Habilitation à Diriger des Recherches en sciences : Sam Meyer
La soutenance est publique
Maître de Conférences : Sam Meyer
Laboratoire INSA : Microbiologie, Adaptation et Pathogénie (MAP)
Rapporteurs :
- Macro Cosentino-Lagomarsino
- Bianca Sclavi
- Andrew Travers
Jury :
- Hubert Charles
- Macro Cosentino-Lagomarsino
- Florence Hommais
- Georgi Muskhelishvili
- Bianca Sclavi
- Andrew Travers
Informations complémentaires
-
INSA Lyon - Bibliothèque Marie Curie - Amphi Emilie du Châtelet - Villeurbanne
Derniers évènements
Tous les évènements
Formation
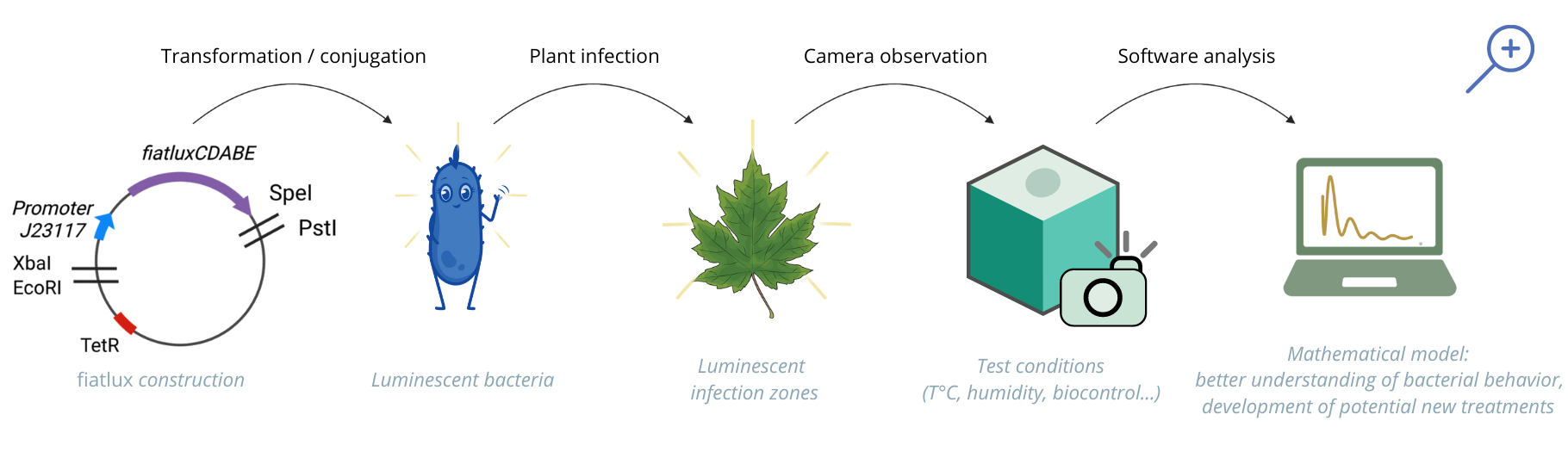
iGEM FIAT LUX : des bactéries phytopathogènes luminescentes
Equipe la plus récompensée lors de la plus grande compétition internationale étudiante en biologie de synthèse, le concours iGEM, FIAT LUX a tout raflé : 2e place générale au concours dans la catégorie undergraduate ; sélectionnée parmi les 7 meilleures équipes sur les 370 participantes ; élue meilleur projet dans le domaine de la nutrition et de l’alimentation ; meilleur hardware ; meilleur assemblage de pièces d’ADN ; meilleure présentation au public ; meilleur site internet et meilleure vulgarisation…
Pourquoi un tel engouement ? Leur nouvel outil de suivi de bactéries phytopathogènes basé sur la luminescence, permet de mieux comprendre les infections de plantes en temps réel sans méthodes intrusives.
Pendant près d’un an, quatorze élèves-ingénieurs du département biosciences de l’INSA Lyon et une étudiante de l’Université Lyon 1, ont travaillé à rendre des bactéries luminescentes afin de visualiser en direct leur développement sur les plantes. Si l’objectif initial était de participer au concours international, au gré des rencontres et des expérimentations, les étudiants sont allés jusqu’à développer un système clé en main : l’outil FIAT LUX.

Annonce de la deuxième place du classement général du concours
dans la catégorie undergraduated (sur 180 équipes) - Paris Expo Porte de Versailles
"Repousser les limites de la biologie de synthèse pour construire des systèmes biologiques capables de faire face aux défis du quotidien" constitue la ligne directrice de la compétition iGEM1. Créée par le MIT, elle rassemble chaque année des étudiants du monde entier pour faire avancer la recherche sur les biotechnologies. Lorsque l’équipe lyonnaise se forme en novembre 2021, c'est d'abord pour « participer à la recherche de solutions alternatives et avoir un impact positif sur le monde agricole », introduit Mathilde Cecchi, en 5e année du parcours biochimie et biotechnologies. « Notre projet était de permettre une meilleure compréhension de l’infection des plantes par des bactéries. Nous espérions pouvoir observer ces pathogènes directement grâce à leur luminescence. Pour cela, nous nous sommes appuyés sur le travail précédent d’une équipe qui s’était appliquée à développer un outil de bioluminescence. »
C’est ainsi qu’une partie de l’équipe entame le travail de génétique. À partir d’une pièce d’ADN provenant d’une bactérie vivant dans les fonds marins, le Photorhabdus luminescens, elle réussit à faire briller leurs bactéries phytopathogènes en les transformant par un assemblage de séquences génétiques spécifiques. Les travaux se concentrent principalement sur deux types de bactéries, les E. coli et les Dickeya solani. « Rendre nos bactéries luminescentes a été le plus long du travail. Nous avons dû fragmenter ce qui existait déjà dans la nature pour le reconstruire, avec le risque que les organismes ne soient plus viables. D’ailleurs, c’est ce qui a été le plus difficile : comme nous faisions quelque chose d’inédit, lorsque ça ne fonctionnait pas, nous ne pouvions pas savoir si c’était dû à nos transformations ou aux conditions dans lesquelles nous les effectuions », indique Théo Mathieu, en 4e année de biosciences.
Ainsi, les étudiants se transforment en apprentis chercheurs, construisant, déconstruisant, reconstruisant. Jusqu’au moment, un jour d’été : « par miracle, nous avions parmi nos colonies de bactéries transformées, une seule qui faisait vraiment beaucoup plus de lumière. On l’a prise en photo avec un smartphone en exposition longue et on a pu observer son comportement en suivant les traces dessinées par sa lumière. Il ne nous restait plus qu’à peaufiner l’outil génétique pour être sûr que la luminescence ne modifiait pas son comportement initial », ajoute Mathilde.
En parallèle, une autre partie de l’équipe est chargée de comprendre le contexte dans lequel la future innovation peut évoluer. « Nous voulions nous inscrire dans la réalité et vérifier comment notre projet allait impacter le monde agricole. Pour que les professionnels soient en mesure de s’emparer de nos outils, nous sommes allés à la rencontre d’acteurs de la filière. Nous avons ainsi compris les besoins et les challenges des agriculteurs, rencontrés des entreprises agro-alimentaires, le ministère de l’agriculture... Plus on avançait dans ces rencontres, plus elles se spécifiaient sur la bactérie Dickeya solani, qui attaque les tubercules de pommes de terre. C’est celle que nous avons utilisée pour notre preuve de concept », explique Élise Piette.
Sur les paillasses du laboratoire MAP2 mises à disposition de l’équipe, les échanges entre étudiants, techniciens et chercheurs élargissent les contours du projet. D’abord avec la conception d’un équipement pour observer le comportement des bactéries, puis la création d’un logiciel d’analyse d’images. « Une fois l’outil génétique fonctionnel, nous avons dû concevoir un hardware pour pouvoir observer le comportement des bactéries luminescentes. Concrètement, c’est une petite chambre noire couplée à une caméra pilotée qui permet de récolter des données précises », ajoute Madeleine Bourseul. « Puis, en discutant avec les biologistes du labo, nous avons remarqué que les possibilités ouvertes par la luminescence n’étaient pas forcément connues. Il n’existait pas de logiciel d’analyse de la luminescence simple, alors nous avons décidé d’en créer un. Nous l’avons imaginé pour les chercheurs avec qui nous étions en contact mais il est aujourd’hui en open source et facilement améliorable », ajoute l’étudiante.
L’open source, c’est le principe qui anime le challenge iGEM. La connaissance créée par les étudiants de l’INSA Lyon et de l’Université Lyon 1 est ainsi accessible aux prochaines équipes et à tous les chercheurs du monde. Pour l’heure, les quinze étudiants ne comptent pas s’arrêter là. Si certains membres poursuivent l’aventure en stage pour en faire un protocole standardisé, tous ont envie d’étendre le champ des possibles. « Nous avons envie de voir si les travaux peuvent aller plus loin, avec une publication scientifique notamment. Nous voudrions faire en sorte que les retombées du projet dépassent le cadre du concours, pour aller sur le terrain », confie l’équipe.
Au terme de cette année d’aventures scientifiques, les étudiants ne retiennent pas seulement leurs capacités à réaliser des prouesses techniques, mais l’importance de la curiosité et du partage. « Nous avons su aller chercher de l’aide un peu partout et s’auto-former sur des sujets très précis selon nos envies personnelles. Par exemple, nous avons reçu de l’aide d’un étudiant de génie mécanique pour construire notre hardware ; nous avons été coachés par le responsable de la section théâtre-études ; et notre contenu graphique a même été réalisé par une diplômée de l’INSA Lyon ! », ajoute Théo. « Au-delà de la magie de cette synergie pluridisciplinaire, il y a un enrichissement personnel qui dépasse toute récompense : le partage des sciences et la richesse des rencontres », conclut Madeleine.

L’équipe FIAT LUX au complet
Les membres de l’équipe :
▪️ Étudiants :
INSA Lyon : Anaëlle Roman, Basma Makhloufi, Elise Piette, Guillaume Fulconis, Laia Darne Clavaron, Léa Le Large, Madeleine Bourseul, Manon Aubert, Marianne Richaud, Marine Aurrand, Mathilde Cecchi, Solène Favier , Théo Mathieu, Tom Roblin.
Université Lyon 1 : Cassie Kohut.
▪️ Encadrants : Agnès Rodrigue (Maître de conférences INSA Lyon, laboratoire MAP) et Erwan Gueguen (Maître de conférences Université Lyon 1, laboratoire MAP)
▪️ Superviseurs : Typhaine Brual (Doctorante INSA Lyon, laboratoire MAP), Marianne Chouteau (Enseignante-Chercheuse INSA Lyon, Directrice adjointe du laboratoire S2HEP), Marie-Pierre Escudié, Marion Létisse (Maître de conférence INSA Lyon), Makram Mghezzi-Habellah (Doctorant ENS Lyon - Laboratoire LBMC).
Contact : igem.insalyon2022@gmail.com
[1] International Genetically Engineered Machine Competition
[2] Microbiologie Adaptation Pathogénie (INSA Lyon/CNRS/Lyon 1/UdL)

Recherche
Des sources chaudes de Yellowstone au test PCR
Trois lettres et plusieurs milliers de tests réalisés par jour en France : « PCR ». Méthode d’amplification de l’ADN largement utilisée par les biologistes, la Polymerase Chain Reaction est seulement connue depuis une trentaine d'années. Plus surprenant encore, c’est grâce à une bactérie récoltée dans le fond des « Hot Springs » de Yellowstone aux États-Unis que la réaction biologique permet de détecter des virus tels que le SARS-CoV-2. Yvan Rahbé, chercheur en microbiologie au laboratoire MAP1 explique le principe de cette technique qui a révolutionné la biologie et permis de répondre rapidement à la crise pandémique.
Yvan Rahbé, chercheur en microbiologie au laboratoire MAP1 explique le principe de cette technique qui a révolutionné la biologie et permis de répondre rapidement à la crise pandémique.
Que se cache-t-il derrière l’acronyme « PCR » et comment a-t-on découvert cette technique ?
La Polymerase Chain Reaction est une technique qui a fait ses premières apparitions dans les années soixante-dix et qui a révolutionné la pratique de la biologie presque autant que les techniques de séquençage massifs de l’ADN dans les années 2000. C’est un procédé permettant aux chercheurs d’amplifier le nombre de copies de fragments génomiques dans un échantillon. Grâce à une enzyme provenant de la bactérie Thermus aquaticus, la « Taq-polymérase », la PCR multiplie les séquences d’ADN.
Pour la petite histoire, c’est dans les profondeurs des sources chaudes de Yellowstone que cette enzyme a été découverte. Capables de résister à des températures et pressions hors-normes, autour de 100°C, ces micro-organismes se sont avérés beaucoup plus résistants que tous les polymérases alors procurables à ce moment-là. Le brevet du test PCR utilisant la Taq-polymérase a été publié en octobre 1990 par Kary Mullis, prix Nobel de chimie (1993). C’est donc grâce à ces chercheurs curieux et un peu casse-cous, que la PCR permet désormais d’identifier la présence de virus très connus : VIH, Cytomégalovirus chez la femme enceinte, hépatites, etc. Elle peut aussi être très utile en archéologie ou en criminologie et plus généralement, dans tous les domaines nécessitant d’étudier les empreintes génétiques.
À l’heure actuelle, le « RT-PCR » est le test de référence pour détecter la présence du virus SARS-CoV-2 dans l’organisme d’un individu à partir d’un prélèvement naso-pharyngé. Comment ce test révèle-t-il la présence du virus ?
Pour pouvoir confirmer ou infirmer la présence d’un virus, les analyses en laboratoires s’appuient effectivement sur la PCR. Ce qu’il faut comprendre, c’est qu’à l’échelle moléculaire, l’analyse est presque impossible sans amplification par PCR car les quantités d’ADN sont insuffisantes pour être étudiées dans un petit échantillon de frottis. Pour le SARS-CoV-2, on cherche à détecter le génome qui lui est spécifique à travers trois étapes. La première consiste à dénaturer l’ADN par un processus de chauffe : formé de deux brins, l’ADN se sépare ainsi en simple brin. Puis, on utilise des petits fragments d’ADN synthétique, les amorces, ces fameux « réactifs » dont on a entendu parler dans les médias, et qui viennent se coller sur l’ADN de l’échantillon : c’est la phase de l’hybridation. La troisième et dernière étape est celle de l’élongation : on utilise la Taq-polymérase, une enzyme capable de reconstituer la séquence d’ADN du gène grâce aux amorces : à la fin du cycle, il y a deux fois plus d’ADN du gène recherché qu’au début. On va ensuite répéter ce cycle plusieurs fois pour obtenir plusieurs copies des gènes propres au SARS-CoV-2. On répète le nombre de cycle autant de fois que nécessaire pour pouvoir dire que le test est positif. Si au bout du nombre de répétitions de cycle fixé par « la valeur-seuil », le test ne révèle toujours pas les gènes spécifiques au virus, on peut dire que le test est négatif.
Pourquoi parle-t-on de « faux-négatifs » ou de « faux-positifs » ? Cela signifie-t-il que les tests RT-PCR ne sont pas fiables à 100% ?
L’efficacité d’un test est d’abord influencée par la valeur-seuil dont je parlais plus haut, qui détermine la positivité du test. Mais ce qui détermine sa performance, comme pour toute méthode d’analyse, ce sont sa « sensibilité » et sa « spécificité », c’est-à-dire la proportion de patients malades que le test détecte correctement (les vrais positifs) et la proportion de patients qui n’ont pas la maladie recherchée dont le test est négatif (les vrais négatifs). Les prérequis définissant ces deux facteurs sont fixés par les CNR, les Centres Nationaux de Références. Aujourd’hui, parmi tous les types de tests capables de détecter le virus, deux sont communément réalisés pour détecter la Covid-19 : le fameux « RT-PCR » et le test sérologique. Ils ont été choisis pour leurs sensibilités et leurs significations biologiques, différentes : la PCR teste le virus et le test sérologique questionne la réponse immunitaire. Dans tous les cas, l’échantillon prélevé sur une personne n’est que le reflet de son état à un moment T et la pertinence d’un test virologique dépend évidemment du moment auquel il est réalisé. Il faut bien garder en tête que nous étudions ici de la matière biologique, qui évolue. C’est d’ailleurs ici tout l’art de la microbiologie !
 1Microbiologie, Adaptation, Pathogénie (CNRS/Université Claude Bernard Lyon 1/INSA Lyon)
1Microbiologie, Adaptation, Pathogénie (CNRS/Université Claude Bernard Lyon 1/INSA Lyon)Le laboratoire MAP est une unité mixte de recherche associant le CNRS, l'Université Claude Bernard Lyon 1 et l'INSA Lyon autour d’un intérêt commun pour les mécanismes d’adaptation et de pathogénie des microorganismes. Au sein d'un campus fortement dédié à la biologie de l'environnement, le laboratoire développe des approches pluridisciplinaires : biochimie, génétique, biologie moléculaire, imagerie, biophysique ainsi que diverses approches globales -génomique, transcriptomique, protéomique, métabolomique, et modélisation mathématique, pour comprendre les mécanismes d’adaptation phénotypique chez tous ces microorganismes.
Podcasts « Les cœurs audacieux » - Saison 2 / Épisode 3 - 9 février 2022

Recherche
Microbiologiste, il observe l’évolution du coronavirus jour après jour
Philippe Lejeune est spécialiste de la contamination bactérienne de matériels médicaux. Enseignant à l’INSA Lyon, au département Biosciences, il communique régulièrement à son entourage ses observations sur la propagation du virus Covid-19 depuis le premier jour de confinement. Récit.
3,6%. Philippe Lejeune intègre ce dernier chiffre au tableau qu’il a construit depuis le 16 mars dernier. Les chiffres, la base. Dans sa tête, il fait le bilan, après un mois de confinement. De plus de 25%, le pourcentage de progression quotidien du nombre de décès dans les hôpitaux dus au coronavirus est passé à 3,6%. Un signe d’espoir pour ce chercheur en microbiologie, attentif à la moindre évolution de la situation. Selon ses analyses, si les mesures de confinement continuent à être respectées, la tendance à la baisse devrait se poursuivre. Une baisse entamée il y a maintenant trois semaines, depuis le 27 mars dernier où la progression du nombre de décès a chuté de 10 points par rapport à la veille, pour ne plus remonter depuis.
Philippe Lejeune le sait, le confinement a porté ses fruits. Dans la colonne de droite de son fameux tableau, il a inscrit le nombre de décès que la France aurait compté si l’augmentation journalière s’était maintenue à 25% après le 27 mars. Plus de 94 000 personnes auraient perdu la vie. Face à un virus dont on ne connait pas encore tous les mystères. Pour ce spécialiste de la contamination microbienne, la première règle à respecter est de se laver les mains. Avec du savon, pendant 20 secondes sous le robinet d’eau tiède. À chaque fois que cela s’impose. En rentrant des courses, après avoir touché un interrupteur, une poignée de porte, un interphone, une rampe d’escalier… La liste est évidemment interminable et le geste sauveur reste le même. C’était ce qu’il était en train de dire à ses élèves de 3e année du département Biosciences de l’INSA Lyon lorsque la menace du virus commençait à prendre de l’ampleur. En plein travaux pratiques pour manipuler une bactérie de manière stérile, ces étudiants apprenaient les rudiments d’un métier à risque : microbiologiste. Les règles d’or : protéger son environnement, protéger les autres, se protéger soi-même et protéger la manipulation de la bactérie en conditions stériles. Un courant d’air dans la salle de cours et l’alarme pouvait être déclenchée. À moindre mal à l’INSA puisque les bactéries utilisées sont inoffensives mais qu’en est-il en conditions réelles ? C’est toute la prise de conscience que s’attèle à déclencher chez ses étudiants Philippe Lejeune, lui qui a passé six années au plus près du danger, à l’Institut Pasteur, pour ses études postdoctorales. Il était également maître de conférences à Paris VII, avant de postuler à l’INSA Lyon, qu’il a rejoint en 1992.
Dans un an, il « partira à la retraite ».
Pour l’heure, il ne sait pas de quoi demain est fait.
Très scrupuleux, il est le premier à avoir respecté les règles du confinement. Féru de marche, il rêve de ce jour où il pourra de nouveau découvrir des sentiers inconnus et respirer un air pur. En attendant, il sort juste pour faire quelques courses, jusqu’à regretter sa dernière initiative. Dans la file d’attente du supermarché, il attend d’être dans le prochain groupe qui sera autorisé à rentrer. Quand la file s’est mise en marche, il sent l’odeur de la cigarette qu’une personne à plus de dix mètres devant lui fumait en avançant dans la file. En tant que microbiologiste, il ne peut s’empêcher de penser que si cette personne était contaminée, elle contaminerait alors tout son entourage immédiat en exhalant la fumée de sa cigarette. Tout comme une personne présentant une toux a priori banale. À partir de ce moment-là, il n’est plus jamais retourné faire ses courses sans masque.
Pour lui, c’est le deuxième geste qui sauve : porter un masque. Ceux qui auraient pu dormir dans les laboratoires de l’INSA ont été donnés aux hôpitaux dès la pénurie annoncée. Tout comme les gants de protection. Dans les laboratoires, y compris le sien, l’unité mixte de recherche CNRS 5240*, les chercheurs ont dû prendre beaucoup de décisions, et trouver des solutions pour maintenir les travaux en cours pendant leurs absences, pour ne pas perdre des heures de recherche. Lorsqu’on leur a dit que l’INSA fermait ses portes, ils ont été les premiers surpris. Ne plus enseigner, oui, mais ne plus chercher, pourquoi ? Et pour combien de temps ?
Il y a quelques jours, la levée du confinement était annoncée pour le 11 mai prochain. D’autres questionnements se bousculent. Philippe, lui, n’a plus à se préoccuper de ses cours. Il a par chance donné le dernier de son semestre quelques heures avant la fermeture de l’école, permettant à tous ses élèves de lui envoyer leurs rapports depuis. Par contre, il ne sait pas quand il pourra remplir son rôle dans les jury de thèses, dont les soutenances sont reportées. Des thèses avec des sujets qui trouvent écho auprès de son âme de chercheur. Comme la multi-résistance aux antibiotiques, qui tuent chaque année en Europe 25 000 personnes. Et d’autres virus infectieux, comme la grippe, dont les ravages sont communément admis et pourtant si meurtriers, chaque année.
Une chose est sûre pour cet observateur scientifique. Il y a un avant et un après coronavirus. Les gens auront appris à se protéger.
*MAP pour Microbiologie, Adaptation et Pathogénie, laboratoire associant le CNRS, l’Université Claude Bernard Lyon 1 et l’INSA Lyon.