Education
iGEM FIAT LUX : des bactéries phytopathogènes luminescentes
Equipe la plus récompensée lors de la plus grande compétition internationale étudiante en biologie de synthèse, le concours iGEM, FIAT LUX a tout raflé : 2e place générale au concours dans la catégorie undergraduate ; sélectionnée parmi les 7 meilleures équipes sur les 370 participantes ; élue meilleur projet dans le domaine de la nutrition et de l’alimentation ; meilleur hardware ; meilleur assemblage de pièces d’ADN ; meilleure présentation au public ; meilleur site internet et meilleure vulgarisation…
Pourquoi un tel engouement ? Leur nouvel outil de suivi de bactéries phytopathogènes basé sur la luminescence, permet de mieux comprendre les infections de plantes en temps réel sans méthodes intrusives.
Pendant près d’un an, quatorze élèves-ingénieurs du département biosciences de l’INSA Lyon et une étudiante de l’Université Lyon 1, ont travaillé à rendre des bactéries luminescentes afin de visualiser en direct leur développement sur les plantes. Si l’objectif initial était de participer au concours international, au gré des rencontres et des expérimentations, les étudiants sont allés jusqu’à développer un système clé en main : l’outil FIAT LUX.

Annonce de la deuxième place du classement général du concours
dans la catégorie undergraduated (sur 180 équipes) - Paris Expo Porte de Versailles
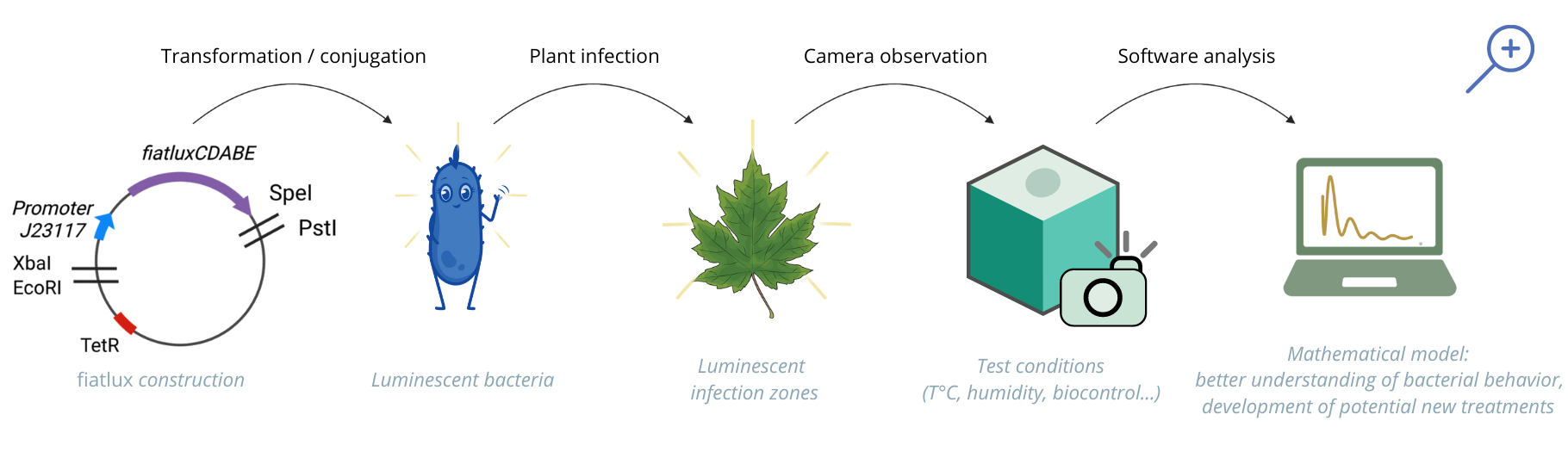
"Repousser les limites de la biologie de synthèse pour construire des systèmes biologiques capables de faire face aux défis du quotidien" constitue la ligne directrice de la compétition iGEM1. Créée par le MIT, elle rassemble chaque année des étudiants du monde entier pour faire avancer la recherche sur les biotechnologies. Lorsque l’équipe lyonnaise se forme en novembre 2021, c'est d'abord pour « participer à la recherche de solutions alternatives et avoir un impact positif sur le monde agricole », introduit Mathilde Cecchi, en 5e année du parcours biochimie et biotechnologies. « Notre projet était de permettre une meilleure compréhension de l’infection des plantes par des bactéries. Nous espérions pouvoir observer ces pathogènes directement grâce à leur luminescence. Pour cela, nous nous sommes appuyés sur le travail précédent d’une équipe qui s’était appliquée à développer un outil de bioluminescence. »
C’est ainsi qu’une partie de l’équipe entame le travail de génétique. À partir d’une pièce d’ADN provenant d’une bactérie vivant dans les fonds marins, le Photorhabdus luminescens, elle réussit à faire briller leurs bactéries phytopathogènes en les transformant par un assemblage de séquences génétiques spécifiques. Les travaux se concentrent principalement sur deux types de bactéries, les E. coli et les Dickeya solani. « Rendre nos bactéries luminescentes a été le plus long du travail. Nous avons dû fragmenter ce qui existait déjà dans la nature pour le reconstruire, avec le risque que les organismes ne soient plus viables. D’ailleurs, c’est ce qui a été le plus difficile : comme nous faisions quelque chose d’inédit, lorsque ça ne fonctionnait pas, nous ne pouvions pas savoir si c’était dû à nos transformations ou aux conditions dans lesquelles nous les effectuions », indique Théo Mathieu, en 4e année de biosciences.
Ainsi, les étudiants se transforment en apprentis chercheurs, construisant, déconstruisant, reconstruisant. Jusqu’au moment, un jour d’été : « par miracle, nous avions parmi nos colonies de bactéries transformées, une seule qui faisait vraiment beaucoup plus de lumière. On l’a prise en photo avec un smartphone en exposition longue et on a pu observer son comportement en suivant les traces dessinées par sa lumière. Il ne nous restait plus qu’à peaufiner l’outil génétique pour être sûr que la luminescence ne modifiait pas son comportement initial », ajoute Mathilde.
En parallèle, une autre partie de l’équipe est chargée de comprendre le contexte dans lequel la future innovation peut évoluer. « Nous voulions nous inscrire dans la réalité et vérifier comment notre projet allait impacter le monde agricole. Pour que les professionnels soient en mesure de s’emparer de nos outils, nous sommes allés à la rencontre d’acteurs de la filière. Nous avons ainsi compris les besoins et les challenges des agriculteurs, rencontrés des entreprises agro-alimentaires, le ministère de l’agriculture... Plus on avançait dans ces rencontres, plus elles se spécifiaient sur la bactérie Dickeya solani, qui attaque les tubercules de pommes de terre. C’est celle que nous avons utilisée pour notre preuve de concept », explique Élise Piette.
Sur les paillasses du laboratoire MAP2 mises à disposition de l’équipe, les échanges entre étudiants, techniciens et chercheurs élargissent les contours du projet. D’abord avec la conception d’un équipement pour observer le comportement des bactéries, puis la création d’un logiciel d’analyse d’images. « Une fois l’outil génétique fonctionnel, nous avons dû concevoir un hardware pour pouvoir observer le comportement des bactéries luminescentes. Concrètement, c’est une petite chambre noire couplée à une caméra pilotée qui permet de récolter des données précises », ajoute Madeleine Bourseul. « Puis, en discutant avec les biologistes du labo, nous avons remarqué que les possibilités ouvertes par la luminescence n’étaient pas forcément connues. Il n’existait pas de logiciel d’analyse de la luminescence simple, alors nous avons décidé d’en créer un. Nous l’avons imaginé pour les chercheurs avec qui nous étions en contact mais il est aujourd’hui en open source et facilement améliorable », ajoute l’étudiante.
L’open source, c’est le principe qui anime le challenge iGEM. La connaissance créée par les étudiants de l’INSA Lyon et de l’Université Lyon 1 est ainsi accessible aux prochaines équipes et à tous les chercheurs du monde. Pour l’heure, les quinze étudiants ne comptent pas s’arrêter là. Si certains membres poursuivent l’aventure en stage pour en faire un protocole standardisé, tous ont envie d’étendre le champ des possibles. « Nous avons envie de voir si les travaux peuvent aller plus loin, avec une publication scientifique notamment. Nous voudrions faire en sorte que les retombées du projet dépassent le cadre du concours, pour aller sur le terrain », confie l’équipe.
Au terme de cette année d’aventures scientifiques, les étudiants ne retiennent pas seulement leurs capacités à réaliser des prouesses techniques, mais l’importance de la curiosité et du partage. « Nous avons su aller chercher de l’aide un peu partout et s’auto-former sur des sujets très précis selon nos envies personnelles. Par exemple, nous avons reçu de l’aide d’un étudiant de génie mécanique pour construire notre hardware ; nous avons été coachés par le responsable de la section théâtre-études ; et notre contenu graphique a même été réalisé par une diplômée de l’INSA Lyon ! », ajoute Théo. « Au-delà de la magie de cette synergie pluridisciplinaire, il y a un enrichissement personnel qui dépasse toute récompense : le partage des sciences et la richesse des rencontres », conclut Madeleine.

L’équipe FIAT LUX au complet
Les membres de l’équipe :
▪️ Étudiants :
INSA Lyon : Anaëlle Roman, Basma Makhloufi, Elise Piette, Guillaume Fulconis, Laia Darne Clavaron, Léa Le Large, Madeleine Bourseul, Manon Aubert, Marianne Richaud, Marine Aurrand, Mathilde Cecchi, Solène Favier , Théo Mathieu, Tom Roblin.
Université Lyon 1 : Cassie Kohut.
▪️ Encadrants : Agnès Rodrigue (Maître de conférences INSA Lyon, laboratoire MAP) et Erwan Gueguen (Maître de conférences Université Lyon 1, laboratoire MAP)
▪️ Superviseurs : Typhaine Brual (Doctorante INSA Lyon, laboratoire MAP), Marianne Chouteau (Enseignante-Chercheuse INSA Lyon, Directrice adjointe du laboratoire S2HEP), Marie-Pierre Escudié, Marion Létisse (Maître de conférence INSA Lyon), Makram Mghezzi-Habellah (Doctorant ENS Lyon - Laboratoire LBMC).
Contact : igem.insalyon2022@gmail.com
[1] International Genetically Engineered Machine Competition
[2] Microbiologie Adaptation Pathogénie (INSA Lyon/CNRS/Lyon 1/UdL)
Keywords (tags)
News list
L’INSA Sailing Club en route vers le plus grand événement sportif étudiant d’Europe